Dear iGEMers
This website is operated in collaboration with iGEM Gifu and iGEM Qdai. This article have compiled information about iGEM in Japanese to provide a new information resource for new Japanese iGEMers in the future.
Let's go iGEM Qdai wiki and iGEM Gifu 2021 wiki !
本記事は、iGEM Japan 輪読会にて、iGEM Gifuによって紹介された、iGEM EPFL 2019 のプロジェクトについて紹介していきます。

[iGEM Japan 輪読会] テーマ : EPFL 2019 by iGEM Gifu
テーマ
チーム : iGEM EPFL 2019
プロジェクト : Toehold switchを用いたファイトプラズマ病検出用バイオセンサー開発
導入
iGEM EPFLが活動しているスイスでは、ワイン醸造用のブドウ栽培が盛んにおこなわれています。現在、ヨーロッパをはじめとしたブドウの栽培地域ではFlavescence Dorée (FD)、Bois Noir (BN)という二つのファイトプラズマ (Phytoplasma) 病が問題となっています。
FDは植物病原菌の一種であるCandidatus Phytoplasma vitisがブドウに感染することで生じるファイトプラズマ病です。FDを発症したブドウの樹木には葉の変色や収縮、果実の萎縮や乾燥といった症状が現れます。
この病気は昆虫によって媒介され、非常に高い感染力を持ちます。FDの感染が発覚した場合、媒介する昆虫を死滅させるため農家は大量の農薬を散布します。また、感染した樹木は直ちに焼却処分されます。仮に農場の20%以上の樹木の感染が発覚した場合は、感染の有無に関わらず全ての樹木を焼却処分する必要があり、農家は甚大な損害を被ることになります。
これに対してBNはCandidatus Phytoplasma solaniの感染によってブドウが発症するファイトプラズマ感染症です。BNを発症したブドウにはFDとほとんど同じ症状が現れますが、感染力はFDに比べて弱いため、農園内で被害が拡大する危険性は低いという特徴があります。そのため、感染した木を処分することで対処が可能です。
FDとBNは発症後にほとんど同じ症状が現れるため、目視で区別することは極めて難しいです。これらを識別できる精密な検査を行うためには専門の機関に依頼する必要があり、結果が判明するまでには3日から15日間の期間を要します。
これらの問題からファイトプラズマ病の症状が確認された場合はFDの感染による被害の拡大を抑えるために、FD、BNの検査を行う前に農薬の散布と焼却処分が行われています。
そこで、EPFLチームは環境に負担が大きい過剰な対応を取っている現状を改善があると考えました。そこでFPFLチームでは農家が自ら検査を行うことができ、結果が即日判明する、迅速で簡単に使用可能な検査キットを作成することを目指しました。
toehold switchについて
実験概要の前にこのプロジェクトでバイオセンサーとして重要な役目を担うtoehold switchについて簡単に説明します。
toehold switchとは、RNAの二次構造を利用した翻訳調節機構のことです。
toehold switchはtoehold領域と呼ばれる配列とその配列に相補的なRNA(trigger RNA)によって下流の遺伝子発現を調節します。toehold領域は下流に存在する配列と部分的に相補となる配列を持っているためステムループ構造を形成します。
このステムループ構造にはリボソーム結合部位(RBS)や開始コドンが内包されます。RBSがステムループ内に格納されるとリボソームの結合が著しく阻害されるため、下流に配置された遺伝子の発現が抑制されます。一方で、trigger RNAが存在する条件下では、toehold領域は自身と完全に相補的な配列であるtrigger RNAと優先的に結合します。
toehold領域とtrigger RNAの結合により、ステムループ構造が解けてRBSや開始コドンが解放されます。これにより、リボソームが結合しやすい状態になり、toehold switch下流の遺伝子発現が促進されます。
toehold領域を任意の配列に設計することにより、特定のtrigger RNA存在下で遺伝子発現を行うサーキットを組み立てることが可能になります。
また、toehold switchはステムループ構造にRBSや開始コドンを内包するものの、toehold領域にRBS( AAGGAG )や開始コドン(AUG)の相補配列を必要としません。
この特徴により、他の翻訳調節機構と比べて塩基の制約が少なくなり、設計の自由度が高いといった利点を持ちます。
toehold switchは遺伝子発現を制御するためのツールとして利用できるだけでなく、toehold領域を標的となるウイルスや細菌に特異的な配列に設計することで、病原体を検出するバイオセンサーとして応用することも可能です。
toehold switchの詳しい説明に関しましては、Greenらの論文に記載してありますので参考にしてください
参考文献 : Green et al., Toehold Switches: De-Novo-Designed Regulators of Gene Expression, 2014
研究概要
このチームが行った実験は、大きく分けて1. DNA抽出、増幅、2. 標的の検出、3. OnePot Pureの三段階に分けられます。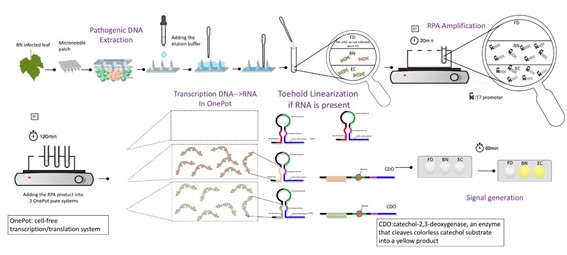
図1 実験のワークフロー
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Design
マイクロニードルをブドウの葉に刺し、これを専用のバッファーで洗い流すことで標的のDNAを抽出します。獲得したDNAを増幅し、Toehold-Switchによるバイオセンサーで検査を行い、感染の有無を確認します。
1a. DNA抽出、増幅
DNA抽出はEPFLチームが3Dプリンタを用いて自作したハードウェアを使用して行われました。マイクロニードルはPVAL (ポリビニルアルコール) を用いて作成されたスタンプ状のもので、ケースに直接シリンジを装着できるように設計されています。シリンジとチューブを装着してバッファーを流し込むことで、マイクロニードルで葉から抽出したDNAを直接回収することが可能になりました。
一般のキットで用いられるスピンカラム法のような複数の作業工程を経ずにDNA抽出を行うことができるため、専門的な知識や高価な機器を必要とせず、簡単に利用できることが示唆されました。
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Hardware
このツールは農地での使用を想定しているため、DNAの増幅を行う際にサーマルサイクラーのような機器を用いずに利用できることを目標としました。
DNAの増幅方法を選択するにあたり、EPFLは以下のような条件を設けました。
1. 等温条件でDNAの増幅が可能である。
2. 一度の反応で複数種のDNA配列を増幅可能である。
3. DNAを迅速に増幅可能である。
上記の条件を元にNucleic Acid Sequence-Based Amplification(NASBA)、Loop Mediated Isothermal Amplification(LAMP)、Recombinase Polymerase Amplification(RPA)の3種の増幅方法による増幅を検討した結果、EPFLはRPA法によるDNA増幅が適しているとして採用しました。
RPAでは、標的配列を囲むFwプライマーとRvプライマー、相同組み換え酵素、一本鎖DNA結合タンパク質、DNAポリメラーゼが機能してDNAを増幅します。また、EPFLはバイオセンサーとしてRNAを標的とする調節機構Toehold Switchを採用したため、Fwプライマーの5‘末端側にはT7プロモーターを付加しました。
RPAは、まずプライマーと相補配列のDNAが相同組み換え酵素によって入れ替わることで開始されます。これにより二本鎖DNA中に相補性を失った一本鎖DNA領域が生じ、ここに一本鎖DNA結合タンパク質が結合することで再会合を防ぎます。そして、DNAポリメラーゼによるDNA合成が進行し新たな鎖が合成されます。
この反応を繰り返すことでDNAは増幅されます。RPA法の詳しい説明はこちらのNEBのサイトを参考にしてください。
参考文献 : NEBのサイト
FDを検出するための標的配列には、FDを引き起こすC. Phytoplasma vitisの16s rRNA遺伝子のR16F2n/R16R2 (F2N/R2) 内の配列が選択しました。
選定されたF2N/R2N内の標的配列はC. phytoplasma vitis間で保存されています。さらに、誤検出を抑えるため、BNを生じさせるC. phytoplasma solaniの16s rRNA上の配列とは相同性が低い配列が選択されました。BNを引き起こすC. phytoplasma solani の標的配列とは異なる16S rRNAをコードする領域を選択しました。
BNの標的配列はC. phytoplasma solani に特異的なゲノム配列領域から選択されました(GenBank accession number AF447593)。
内部コントロール(Endogenous Control, EC)の標的配列はヨーロッパブドウの葉緑体ゲノムから得られた配列を選択されました。ECはブドウの葉からのDNA抽出や等温増幅が確実に行われたのかを確認するために必要になります。
実際の検査キットでECが検出できなければDNAの抽出、あるいは増幅が正確に行われていなかったことが分かるため、誤って陰性と判定される危険性を抑えることが出来ます。
RPAによりFDを検出するためのDNAの増幅がどの程度行われるのか検証したところ、DNA濃度は10コピー、100コピーという低濃度でも検出可能であることが判明しました(図3 a)。BN検出DNAに関しては検出限界を評価できませんでした。加えて、複数種のDNA断片を同時に増幅させた際も正常に増幅されたことが示唆されました(図3 b)。
さらに、ブドウ抽出物中のフェノール等が増幅を妨げる可能性を考慮して、ブドウ抽出物が反応液中に存在する状態でもDNA増幅が可能か検証しました。
結果、RPAが正常に機能しFDに関しては検出限界も10コピーであることが分かりました(図3 c)。
a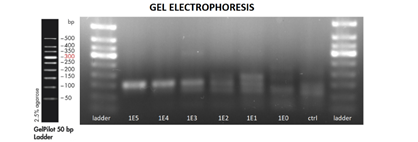
b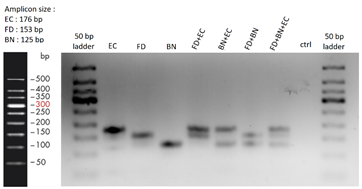
c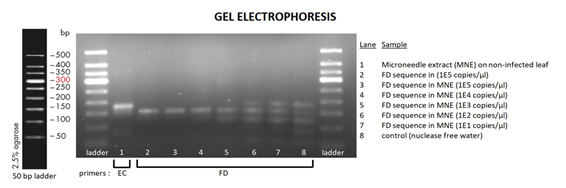
図3 a:FD検出遺伝子の濃度別増幅後アガロースゲル電気泳動、b:複数遺伝子の同時増幅後のアガロースゲル電気泳動、c:ブドウ抽出液を含むサンプルでのDNA増幅産物のアガロースゲル電気泳動
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Amplification
1b. ブドウの葉から抽出したDNAの増幅、発現
実際に病気に感染したブドウの葉からキットを用いてDNA検出を行いました。内部コントロール、FDともにDNAが増幅されていることが確認されました(図4 a, b)。次にこれらのDNAアンプリコンが転写可能であるのかをHiScribe™ T7 Quick High Yield RNA Synthesis Kitを使用してin vitroで確認したところ、内部コントロール、FD、BNのすべてのアンプリコンにおいて充分に転写が行われていました(図4 c)。
a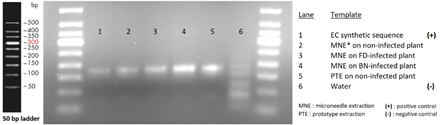
b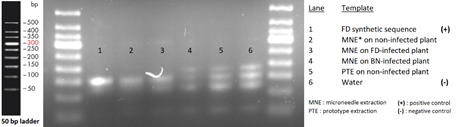
c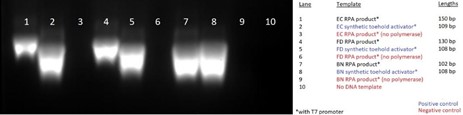
図4 a:合成ECとブドウの葉からマイクロニードル抽出したDNAの増幅後アガロースゲル電気泳動 b:合成FDとブドウの葉からマイクロニードル抽出したDNAの増幅後アガロースゲル電気泳動 c:RPA転写産物のアガロースゲル電気泳動
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Amplification
2. 検出
シグナル検出にはtoehold switchを利用しました。また、EPFLではtriggerによって発現する遺伝子としてカテコールの酸化反応を触媒するカテコール-2,3-デヒドロゲナーゼを選択しました。この酵素によってカテコールは2ヒドロキシムコン酸セミアルデヒドに変換され無色のカテコールから黄色に変化します。
設計した検査キットは屋外での使用が想定されており、呈色反応を選択することで、白昼のような強い光の元でも簡単に陰性と陽性を容易に判定出来るようにするためです。trigger RNAはFD、BN、EC由来のDNAを転写して得られたものです。
toehold領域、RBS、開始コドン、リンカーによりtoehold switchは構成されています。目的のtoehold switchを得るために、設計したDNA配列を含むプラスミドを作成し、形質転換して発現させました。このとき、プラスミドが転写されてToehold-Switchが生じたことを確認するためにカテコール-2,3-デヒドロゲナーゼの代わりにレポーター遺伝子としてGFPを用いました。
設計したtoehold switchの性能を確かめるため、dsDNAの転写産物とssDNAをトリガーとしてシグナルを検出する実験を行いました。ssDNAはトリガーとして機能し、dsDNAと異なり転写等の要素に影響されないため、toehold switchが機能しているのかを確認するのに適しています。これらを比較すると転写産物であっても十分にトリガーとして機能することが分かりました。ssDNAについては、100nMであってもtoehold switchが機能することが分かりました。
a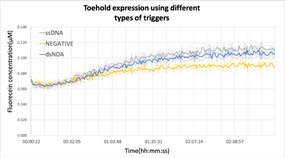
b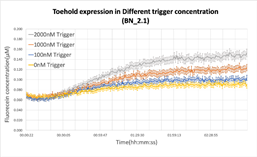
図5 a:ssDNA、dsDNAによるToehold-Switch発現に伴う蛍光強度測定 b:トリガー濃度別toehold switch発現に伴う蛍光強度測定
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Detection
次に実際にtoehold switchの下流にカテコール酸化酵酵素を配置しtoehold switchによって発現可能かcell-free systemを用いて検証しました。30分で呈色が確認され、45分、60分、90分と経つにつれ色が濃くなりました。これにより、toehold switchとcell-free system によってカテコール酸化酵素が発現可能であることが示唆されました。
図6 Toehold-Switch発現によるカテコールの酸化に伴う呈色反応
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/Results
3. OnePot Pure
これらのtoehold switchとCell-free systemを用いて、レポータータンパク質の転写、翻訳を行いました。Call-free systemはin vitroでの転写、翻訳が可能にするツールです。中でもPure systemは転写、翻訳に必要な因子のみで構成されたCell-free、夾雑物をほとんど含まないため、精製を前提としたタンパク質の合成などに利用されます。
一方で、市販のキットを使用すると実験に運用コストが高いことが難点です。EPFLではPure systemを自作することで安易かつ安価にタンパク質を発現させようと考えました。
本体のPure systemでは転写、翻訳に必要な遺伝子のクローニングや精製を別々で行っているため、製造には時間とコストが掛かります。そこでEPFLでは個々の遺伝子を少量ずつ大腸菌でクローニングし、最終的に一つの容器に併せて大量培養、同時に精製することで必要な時間と経費を抑えるOnePot Pureを考案しました。遺伝子をOnePot Pureは大きく分けて翻訳因子アミノアシルtRNAシンターゼ、リン酸基転移酵素に分類されるタンパク質36種から構成されます。
OnePot Pureに必要なタンパク質は、96ウェルマイクロプレートで目的タンパク質遺伝子を含む組み換えプラスミドで形質転換した大腸菌を少量培養したのち、別の大きなフラスコに集め大量培養することで合成しました。
その後遠心分離で集菌、バッファーで懸濁、夾雑物を除去しニッケルカラムで精製しタンパク質を集めました。
リボソームを集める際には大腸菌を少量培養後に大量培養を行い、集菌、溶菌、二段階遠心分離により夾雑物を除去します。その後疎水性相互作用クロマトグラフィーで精製し、スクロースバッファーを加えて超遠心をかけて精製します。最後にフィルターで濃縮し数回分の反応に使用可能なリボソームを得ます。
このような方法で得られた精製後のタンパク質をSDS-PAGEで同時に確認しました。これは個々のタンパク質の機能を確認しているわけではなくマーカーのサイズから予想されるタンパク質の発現を確認しているものです。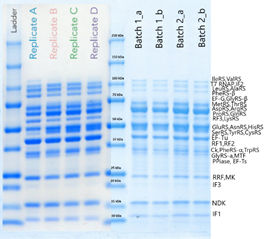
図7 OnePot Pureに必要なタンパク質精製後のSDS-PAGE
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/OnePot_Pure
得られたタンパク質を用いて作成したOnePot Pureと市販のPure system、それぞれのネガティブコントロールを比較したところ、自作したOnePot Pureで発現したタンパク質が最も多いという結果が得られました。
DNAのテンプレート量を変化させても同様の結果が得られました。サンプルとして使用したDNA溶液の濃度と得られたタンパク質の量を比較した結果から、OnePot Pure を用いた反応に最適なDNA量は5nMであるとしています。
a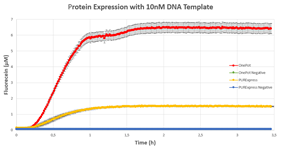
b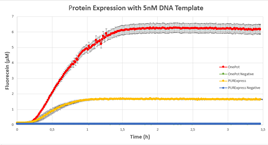
c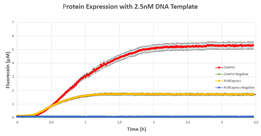
d
図8 a, b, c:各DNA濃度のOnePot Pure、Pure system、コントロールで発現したGFPの蛍光濃度 d:DNA濃度ごとの蛍光物質発現量の比較
Team:EPFL-2019, 2019, ViTEST, https://2019.igem.org/Team:EPFL/OnePot_Pure
まとめ
実験室レベルでの実験では、ブドウがFD及びBNを発症させるファイトプラズマに感染しているのか否かを判別することができるツールの設計と、各反応段階における結果を得ることができました。
これらの要素を組み合わせることで迅速なFD検出ツールの作成が可能になることが予想されます。
一方で、抽出、増幅、検出の一連の操作を連続して検証し、検出可能であると結論づけられたわけでありません。
現場で利用可能な検査方法として確立されるには一連の操作を通して検出が可能と証明される必要があり、実際に検査キットとして利用されるためには再現性の確認、検査精度の検証などの追加実験が必要不可欠です。
試作キットを使用した反応が、検査として信頼性の高いデータを安定的に示すような実証実験を繰り返すことで、実用化への道が開かれると予想されます。
EPFL2019は2019のGrand Prizeを獲得しており、さらに2つのAwardを受賞、3つのAwardにノミネートされている非常に完成度の高いプロジェクトです。
特にウイルスや細菌の核酸を標的としたバイオセンサーを設計をするチームには参考になる点が多いです。プロジェクトの内容だけでなくWikiやプレゼンの構成など、非常に参考になる点が多いチームです。是非、一度WikiやYouTubeのプレゼンなどを視聴してみてください。