今回より輪読会の参加チームのローテーションによる発表が始まりました。第3回の発表を行ったのは、九州大学に拠点を置くiGEM Qdaiです。
本記事は輪読会でのiGEM Qdaiによる発表の内容をもとに構成されています。

「RNAiのiGEMにおける応用」- iGEM Japan 2022 定例会 (輪読会) #3
目次
始めに
今回の輪読会のテーマは「iGEMにおけるRNAiの応用方法」で、以下の2つのプロジェクトが紹介されました。
- SZU-China 2019 Micrancide
- Bielefeld-CeBiTec 2018 nanoFactory
参考:
RNAiとは?
RNAiは、細胞内に、標的のmRNAと相補的な低分子RNAが存在する時に、標的のmRNAに、mRNAを分解する酵素が誘導されることにより、mRNAが分解され、それによって遺伝子の発現が抑制される現象です。
RNAiにおいて重要な役割を果たす要素
siRNA
標的となるmRNAへと分解酵素を誘導する役割と果たします。
ダイサー
RNA分解酵素の一種であるダイサーは細胞内へと低分子RNAを導入する働きをもつベクターであるdsRNAやpre-miRNAをそれぞれsiRNAやmiRNAに分解します。
RISC
RISCの正式な名称はRNA誘導サイレンシング複合体で、低分子RNAと分解酵素の一種であるアルゴノートタンパク質から構成されるヌクレアーぜ複合体です。このRISCの触媒部分が低分子RNAの誘導のもと、標的mRNAを分解します。
RNAiの作用機序
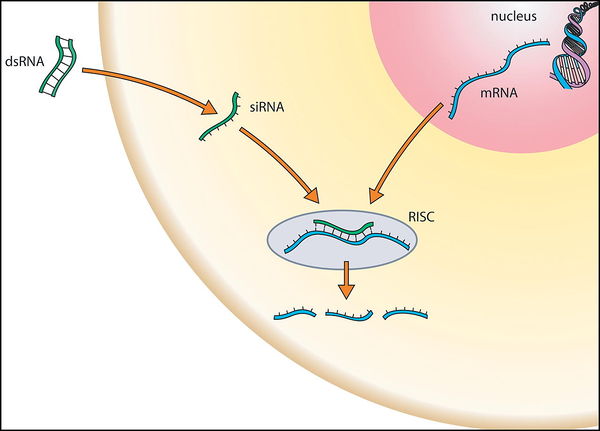
RNAiの作用機序
出典 : 脳科学辞典 RNA干渉 https://bsd.neuroinf.jp/wiki/RNA干渉
1. 細胞内へ外部からdsRNAなどのsiRNAのベクターを導入すると、まずダイサーが働き、ベクターをsiRNAへと分解します。
2. siRNAがRISCタンパク質(アルゴノート)と結合することによってRISCを形成します。
3. 低分子RNAの誘導のもと、標的mRNAとRISCが特異的に結合すると、RISC自身のもつヌクレアーぜによって破壊されます。
以上がRNAiの大まかな流れです。
SZU-China 2019
一つ目のプロジェクトです。このプロジェクトは、侵略的外来種として世界中で生態系に大きな悪影響を与えている植物であるツルヒヨドリに対する除草剤をRNAiを用いることで作成しようというものです。
ツルヒヨドリとは?
ツルヒヨドリは世界ワースト10の雑草とされている熱帯原生の蔓性植物です。
SZU-Chinaのキャンパス内に繁茂するツルヒヨドリ
出典 : iGEM2019 SZU-China Micrancide Description https://2019.igem.org/Team:SZU-China/Description
特徴
- 成長速度が速く、繁殖力が高い。
- 新芽には他の植物の成長などを抑制する他感作用を引き起こす物質を分泌する能力がある。
- 他の植物の上を覆うように広がることにより、日光を遮り枯らす。
- 温暖化の影響により、生息域が拡大している
SZU-Chinaによる従来の駆除方法の検討
物理的駆除
機械や人の手によって抜く、刈る方法。物理的に引っこ抜くのが駆除に最良の方法であるという一方で、間違って違う植物を引っこ抜いてしまうなどのリスクが存在します。
化学的駆除
化学的な除草剤を用いる方法ではツルヒヨドリの密集度が高い場合に非常に効果的である一方、有害物質の残留などによる環境への影響が懸念されます。
生物的駆除
生物的防除の方法は、持続的な管理方法として最適であるとこのチームは述べていましたが、外来生物を連れてくることによる生態系への影響は計り知れず、リスクが高いです。
SZU-Chinaによる提案
そんな中、SZU-ChinaはRNAiに目をつけました。
具体的には、
- CRISPRなどを用いた遺伝子改変技術とは異なり、遺伝子発現の永続的な変化を引き起こさない。
- RNAは他の除草剤に用いられる化学物質よりも不安定である。
- ターゲットであるツルヒヨドリ以外には害を及ぼさない
という点からRNAiを利用した除草剤を作成しようとプロジェクトを立案しました。
プロジェクトの流れ
①ターゲット遺伝子の決定
初めにターゲットとする遺伝子を決定し、それに対するsiRNAの配列の決定が行われました。初めは植物内でよく発現する呼吸関連の遺伝子をターゲットとしていましたが、途中からは実験する際に目で見て除草剤の効果が確認できるようにクロロフィル関係の遺伝子をターゲットとしました。
siRNAの配列は、ツルヒヨドリの葉から抽出して得た、Total RNA (全長RNA)を、RNA-seqで解析することで決定されました。その際、オフターゲットの確認も行い、ツルヒヨドリにユニークな遺伝子を選択しました。
②siRNAを細胞内へ届けるベクターの決定
次に細胞内へsiRNAを届けるベクターの決定が行われました。SZU-Chinaは複数のベクターを検討した結果、SZU-Chinaは、shRNAベクターをベクターとして選びました。
③プラスミドの構築
SZU-Chinaが構築したプラスミドには、
- ターゲット遺伝子に対するshRNAを生産する領域
- 環境が酸性となると、細胞の自死を引き起こすself-cracking Mechanism
が組み込まれたようです。
残念ながら、SZU-Chinaがwikiに記載していたプラスミドの設計図は不明瞭であり、判別不能でした。
④shRNAの生産
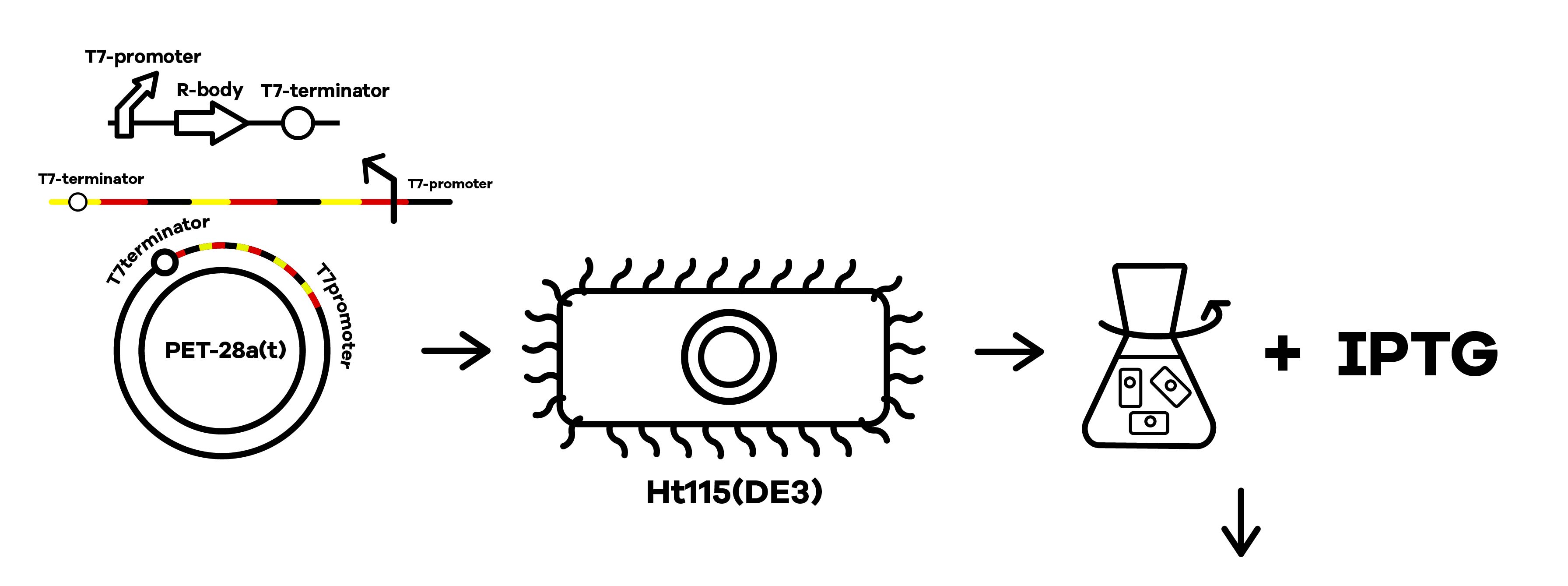
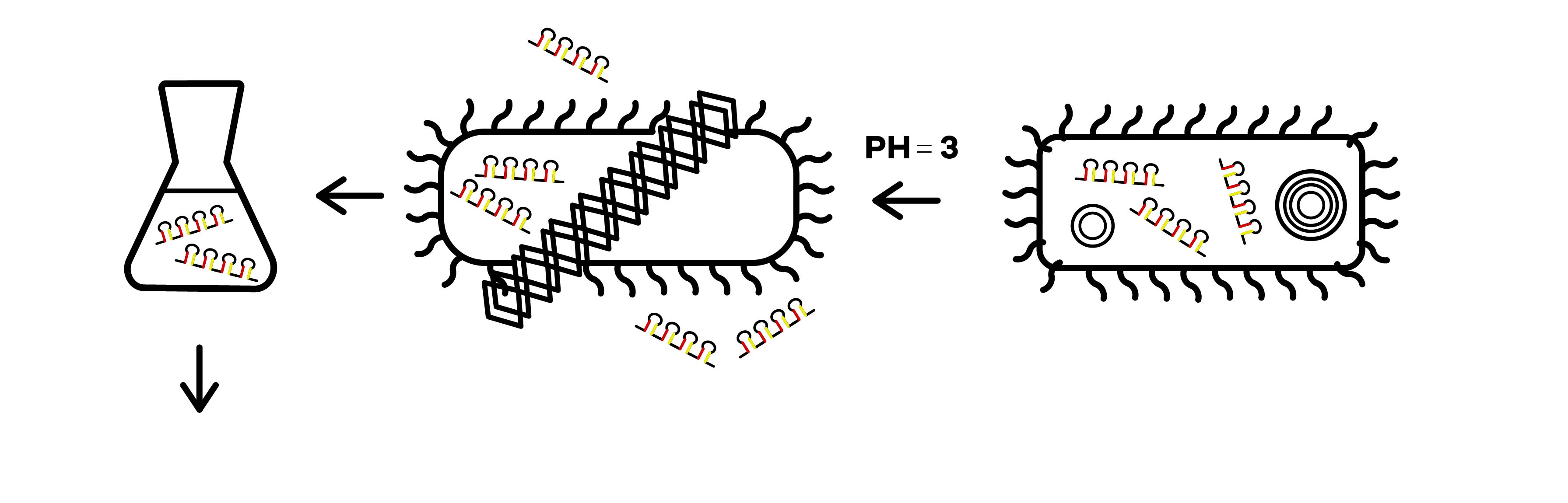
shRNAの生産方法
出典 : iGEM2019 SZU-China Micrancide Design https://2019.igem.org/Team:SZU-China/Design
1. shRNAの生産するプラスミドを大腸菌に形質転換して導入
2. IPTG誘導により、プラスミドの転写を促進し、細胞内でのshRNAの合成を促進
3. 溶液を酸性に傾けることによって大腸菌を死滅させる。
4. 溶液からshRNAを抽出、精製することで除草剤の原料が生産されます。
得られたshRNAを溶かした溶液をツルヒヨドリに噴霧することでRNAiが引き起こされます。
実験による効果の検証
SZU-Chinaが除草剤の効果を測るために
- 効果に関する実験
- 特異性についての実験
- 安定性についての実験
の3つの実験を行いました。
除草剤の効果に関する実験
遺伝子の発現状況を定量的に計測した実験です。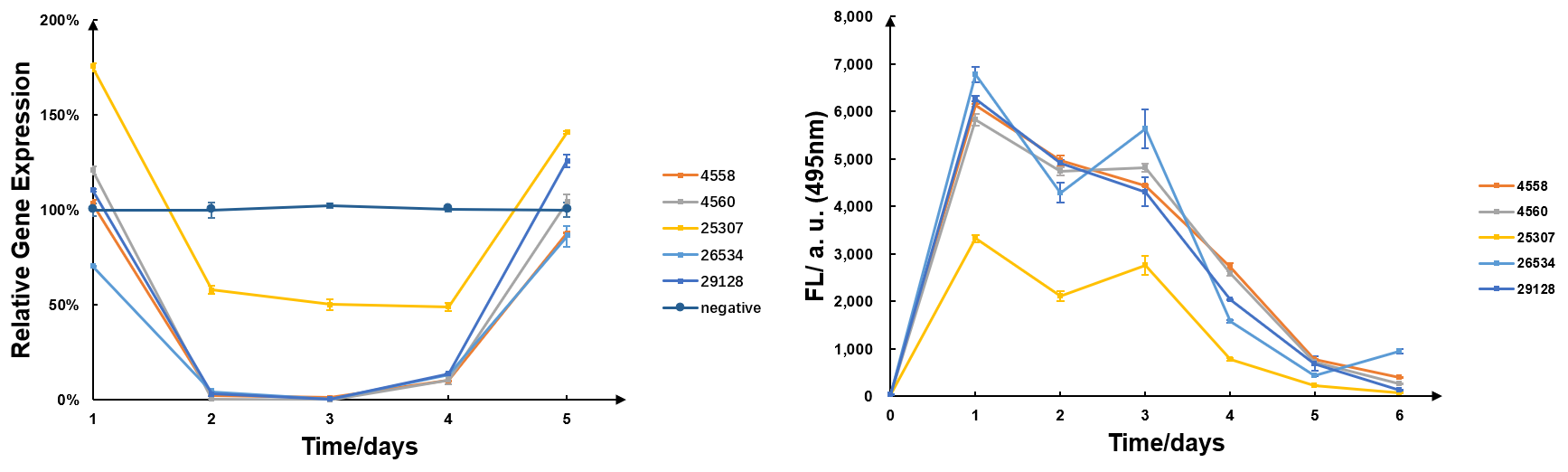
左)遺伝子の発現量 右)蛍光により測定した細胞内のsiRNAの量
出典 : iGEM2019 SZU-China Micrancide Results https://2019.igem.org/Team:SZU-China/Results
図より生体内のsiRNAの量が増加すると、標的遺伝子の発現量が減少し、またその逆の傾向も読み取ることができます。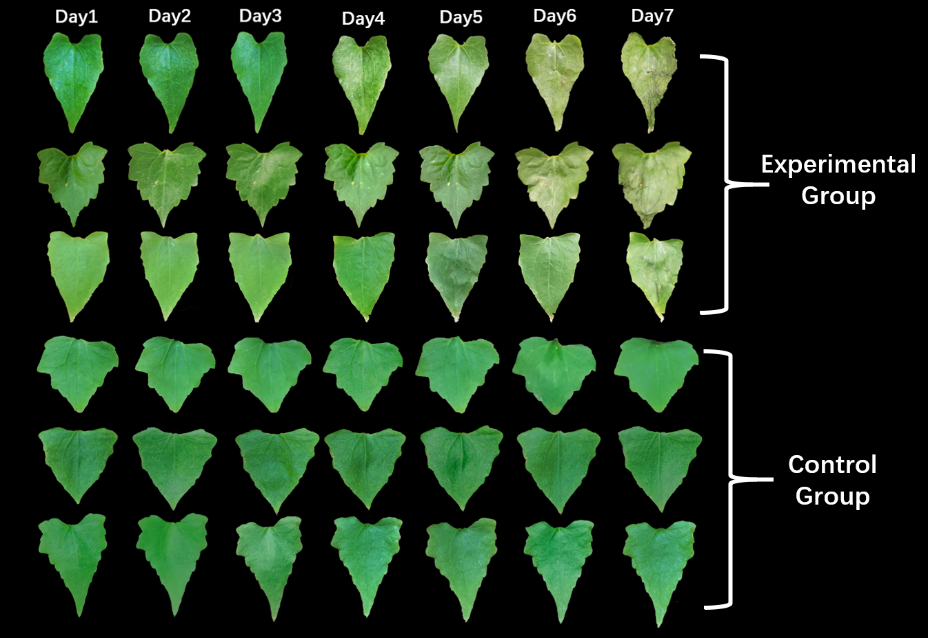
shRNAを7日間毎日噴霧した葉の形態
出典 : iGEM2019 SZU-China Micrancide Results https://2019.igem.org/Team:SZU-China/Results
写真より毎日shRNAを噴霧した方の葉は日が経つに連れて萎れている一方で、純粋な水を噴霧した方の葉は変化がないことがわかります。
除草剤の特異性に関する実験
実験ではシロイヌナズナやタバコといった植物に対してshRNAを噴霧して変化が起きるかどうかを調べています。


上) ツルヒヨドリ, 中) シロイヌナズナ, 下) タバコ
出典 : iGEM2019 SZU-China Micrancide Results https://2019.igem.org/Team:SZU-China/Results
ツルヒヨドリには変化が見られる一方で、他の二つには変化が見られないことがわかります。
除草剤の安定性についての実験
shRNAの安定性についての検証です。SZU-Chinaは除草剤として実際に製品として売り出すことになった時の保管推奨温度を決定しようとしてこの実験を行いました。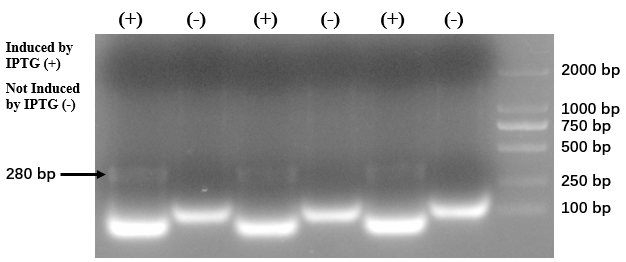
大腸菌から得られたshRNAの電気泳動写真
出典 : iGEM2019 SZU-China Micrancide Results https://2019.igem.org/Team:SZU-China/Results
280bpのバンドがshRNAのものです。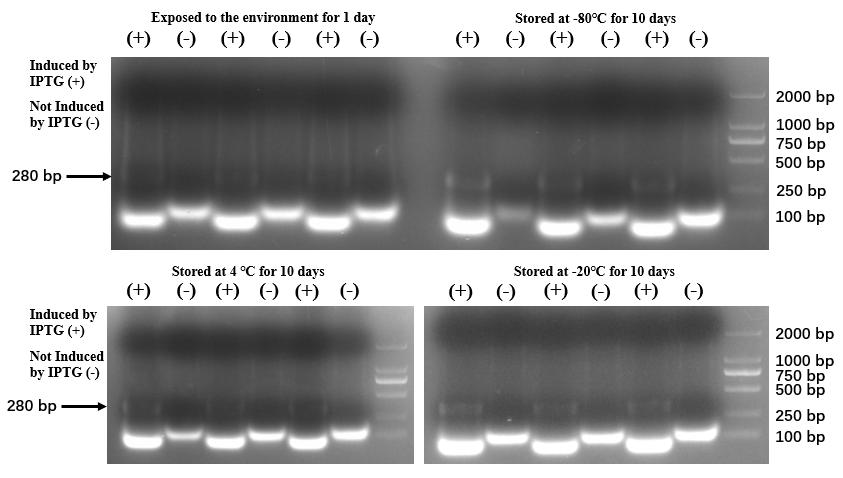
各保存方法の結果
出典 : iGEM2019 SZU-China Micrancide Results https://2019.igem.org/Team:SZU-China/Results
保存方法はそれぞれ、
- (左上) 1日間野外で放置
- (右上) -80℃で10日間保存
- (左下) 4℃で10日間保存
- (右下) 20℃で10日間保存
となっています。
右上の図と右下の図には280bpのバンドを見ることができる一方で、他の二つからは消失してしまっているという結果から、SZU-Chinaは-20℃をこの除草剤の保管推奨温度として設定しました。
まとめ
2019年のプロジェクトとしてSZU-ChinaはRNAiを用いたツルヒヨドリに対する除草剤を作成しました。それは、ツルヒヨドリのクロロフィルに関する遺伝子をターゲットとしたsiRNAを取り込ませることによって、クロロフィルに関する遺伝子の発現を抑えることによって、ツルヒヨドリを枯らしてしますものです。それらは、ツルヒヨドリ以外の植物には影響を及ぼさないものでした。
Bielefeld-CeBiTec 2018 NanoFactory
二つ目のプロジェクトです。このプロジェクトは、2018年にBielefeld-CeBiTecが行った大腸菌を用いて金属を回収しようというものです。このプロジェクトは4つのサブプロジェクト(Toxicity, Accumulation, Silencing, Nanoparticles)により構成されており、今回はその中からSilencingを取り上げられました。
SilencingはCRISPR/Cas9の類似現象を、原核生物である大腸菌において、RNAiを模倣した反応によるノックダウンを行い、再現しようというサブプロジェクトです。
CRISPR/Cas9とは?
関連の深いCRISPR/Cas9の解説です。
CRISPR-Cas9では、標的のDNA配列に対して配列特異的な20塩基程度のガイドRNAを利用してCas9ヌクレアーゼをその標的座位に誘導し、標的となったDNAの二本鎖を切断します。
切断に対し、真核生物は大きく分けて二種類の応答を行います。
1. 染色体上の2つの遊離末端を再度結合させる非相同性末端結合
2. 片方の染色体で損傷が起きた際、もう片方の染色体の相同な部位を鋳型にして修復を行う相同組換え修復。この相同な部位を実験者が用意したDNAに置き換えてしまえば、DNA配列を編集できます。
iGEMにおけるCRISPRの問題点
iGEMには合成生物学を用いた製品の社会実装を目指すという理念があります。その点、CRISPRの特許によるライセンス契約の必要性は、パーツの商用利用においてネックとなってきます。
これを踏まえてBielefeld-CeBiTecはiGEM内で自由に使えるオープンソースなシステムを構築しようと考えました。
大腸菌に備わるsiRNAによる反応の問題点
大腸菌でRNAiを再現する一番大きな問題点は、ほとんどの真核生物とは異なり、細菌の多くはRNAi機構を持っていないということが挙げられます。多くのsiRNAはタンパク質と結合したり、RNAのアンチセンス鎖として働くことによって遺伝子の発現を抑制します。しかしこの手法をそのまま用いると、
- siRNAが再び分離することによって絶対的に発現を抑制することができない
- 翻訳の抑制から無制限の発現へと移行する速度が遅い
という欠点が存在します。そのため、Bielefeld-CeBiTecは大腸菌内で真核生物におけるRNAiを模倣した遺伝子発現制御機構を作成しようと考えました。
改善策
Bielefeld2018による改善案は二種類です。
1. 大腸菌に存在する相同タンパク質であるHfq, RppH, RNase Eを用いてターゲットRNAを分解する機構を作成する。
2. 立体障害による翻訳の阻害を行う。
1の方法
一つ目の方法についてです。このチームは大腸菌でRNAiと同様のメカニズムを確立しつつ、代謝機構への影響を最小限にするために、すでに大腸菌内に豊富に存在するタンパク質を用いて模倣した機構を作ることにしました。
用いられたタンパク質
Hfq
シャペロンの一種で、模倣機構においてsiRNAとターゲットRNAの結合やRppHとの結合を誘導する役割を果たします。
RppH
RNaseEの活性化を行います。
RNaseE
ターゲットRNAの分解を行います。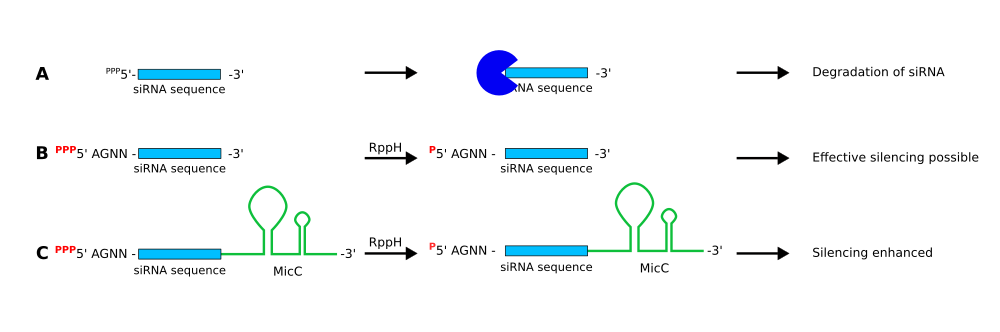
1の方法におけるsiRNAの修飾
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Software https://2018.igem.org/Team:Bielefeld-CeBiTec/Software
(A) のように何も修飾されていないsiRNAは細胞内のヌクレアーゼによって分解されてしまいます。しかし、(B) のようにsiRNAの5末端が一リン酸である場合、RNaseEの触媒活性を増加させ、ターゲットRNAの分解を誘導することができるようになります。また、siRNAの5末端にAGNN配列を付加すると、RNaseEの活性を促進するようです。なお、(C) のように3末端にMicCというスキャフォールドを結合させると、siRNAとターゲットRNAの結合を促進し、またsiRNAの安定性も上昇させるようです。
このように修飾されたsiRNAは、RNAシャペロンHfqによって結合したRppHによって活性化、5末端をピロリン酸から一リン酸へ変換されます。
そして変換されたsiRNAが標的mRNAに結合すると、RNase EによってmRNAが分解されます。
2の方法
次に立体障害による翻訳阻害です。この場合siRNAは安定である方がより良いです。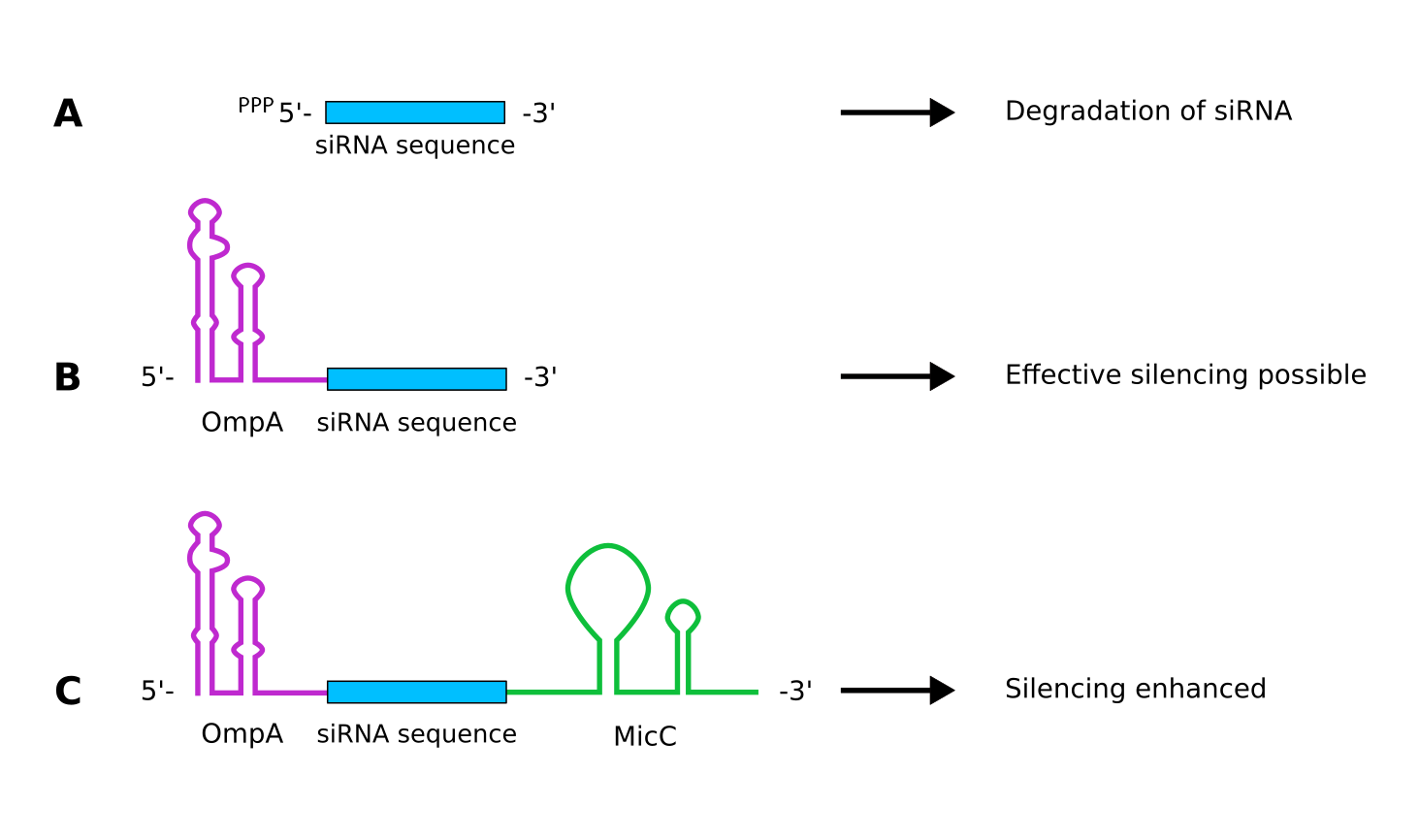
2の方法におけるsiRNAの修飾
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Software http://2018.igem.org/Team:Bielefeld-CeBiTec/Software
wikiによると、5末端にompAを修飾したsiRNAは半減期がかなり長くなるようです。またシャペロンHfqはsiRNAとmRNAの結合を促進しますが、3末端に先ほどと同じMicCを持つものはその効率を最も良くするようです。このようにして作成されたsiRNAはブロッキングによりmRNAの翻訳を阻害します。
システムの検証
この二つの手法による原核生物である大腸菌でRNAiを模倣したシステムをBielefeld-CeBiTecは考案しました。そして彼らはそのシステムが想定通りに働くか、大腸菌内で検証を行うためのシステムTACEを設計、構築しました。
TACEは主にsiRNAを発現するベクターと、ターゲットRNAを発現するベクターに分かれています。
siRNAを発現するベクター
siRNAの発現を行うベクターの方では、発現させたsiRNAを全て比較できるようにすることが重要です。Bielefeld-CeBiTecはそうなるように同じプロモーターを搭載した以下の4つのBioBrickを設計しました。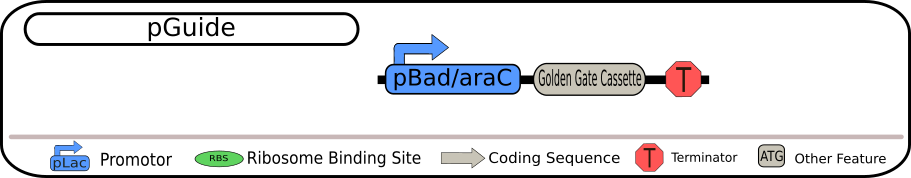
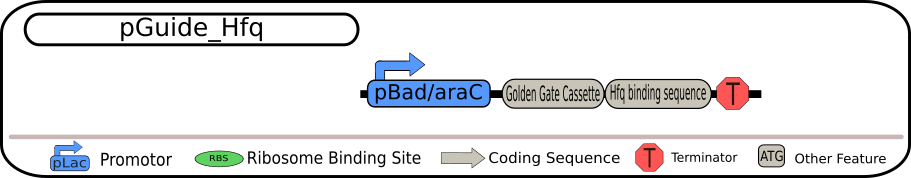
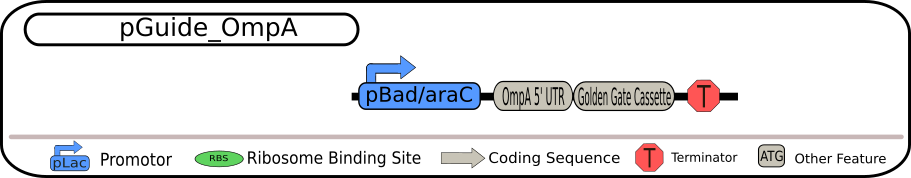
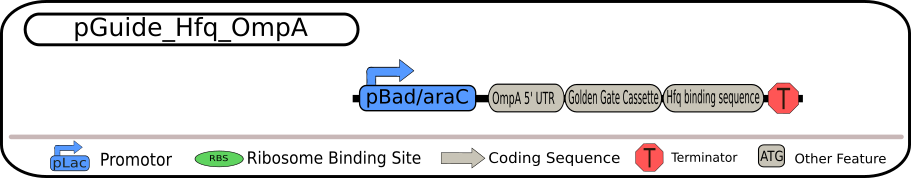
4つのBioBrickの設計図
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Silencing Results http://2018.igem.org/Team:Bielefeld-CeBiTec/siRNA_Results
いずれも制限酵素BbsIを用いて切り出すことができるGolden Gate Assembly (GGA) カセットを含んでいます。そのため、GGAプロトコルを用いることで、GGAカセット全体を特定のsiRNAに置き換えることが可能となっています。siRNAの転写は、strong TerminatorBBa_B0015によって終了され、全ての発現過程が完全に終了されます。
各BioBrickの相違点は、一番上の配列ではそのままsiRNAを転写するようになっていますが、二つ目の配列ではMicC-siRNA由来のHfq結合配列も含まれています。また、三つ目の配列ではsiRNA挿入部位の上流に保護配列としてompA由来の5'-UTRを含み、最後の配列は、二つ目の配列と三つ目の配列の両方の特徴を持つsiRNAを含んでいます。
これらの発現ベクターのうちの1つを、TACEシステムの最初の部分として用いることができます。
ターゲットRNAを発現するベクター
次にターゲットRNAを発現するベクターについてです。このベクターにはpTaleという名前がついており、一つの特定のmRNAを転写します。選択されたRNAは、先ほどのいずれかのsiRNA発現ベクターを使用して作成されたsiRNAによってサイレンシングされる必要があり、Bielefeld-CeFiTecはsiRNAのサイレンシング効果を測定するための最適な条件を得るために、2世代のターゲットベクターを構築しました。
pTale第一世代
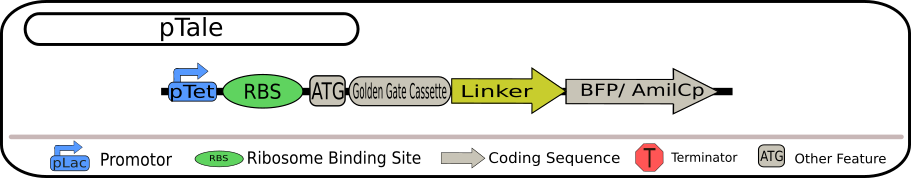
pTaleの設計図
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Silencing Results https://2018.igem.org/Team:Bielefeld-CeBiTec/siRNA_Results
レポータータンパク質としてはクロモプロテインAmilCpと青色蛍光タンパク質BFPの2種類が使われています。ターゲットBioBrickには、GGAカセットとレポータータンパク質の間にリンカーがあることが特徴です。リンカーは立体構造への影響が一般的に軽減されることが知られているために、挿入されたターゲットmRNAは、機能を失うことなくレポータータンパク質と結合を形成すると、Bielefeld-CeBiTecは想定していました。しかし、その相互作用については絶対的なことではないため、それを確認するためにpTale2の設計も行っています。
また、mRNAが破壊された場合、Bielefeld-CeBiTecは、レポータータンパク質は形成されないため、第一世代のpTaleを使用した場合、測定可能な発光はサイレンシング効果により減少すると主張していますが、siRNAがターゲット配列のどこと相補的に結合するか、はっきりとwikiに書かれていなかったため、疑問が残ります。
pTale第二世代
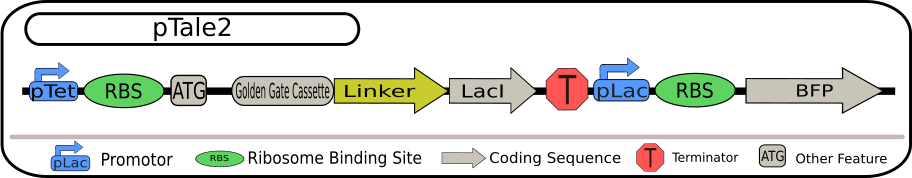
pTale第二世代の設計図
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Silencing Results https://2018.igem.org/Team:Bielefeld-CeBiTec/siRNA_Results
第二世代のターゲットベクターは、ターゲット遺伝子とレポーター遺伝子が直接結合しない形となっており、レポーターがLacプロモーターの後ろにクローニングされる形となっています。ターゲット配列はLacリプレッサーのコーディング領域に融合しているという構造となっているため、ターゲットRNAがサイレンシングされると、測定可能な蛍光が増加することとなります。さらに、Lacプロモーターは誘導および抑制することができるため、TACEシステムに新たに別の制御軸を導入することが可能になっています。
第一世代との大きな違いは、サイレンシング効果が高くなると蛍光が減少するのではなく、増加するように設計されている点です。この構造により、非機能的なレポータータンパク質によって生成される偽陽性を防ぐことができようになっています。
実験
Bielefeld-CeBiTecが行った実験です。彼らは検証システムTACEのベクターの構築に時間がかかってしまい、実験を時間かけて行うことができなかったようです。そんな中でも、ターゲット遺伝子を緑色蛍光タンパク質GFPとしたpTale第一世代のベクターを構築し、検証実験を行いました。検証の方法としてはOD600とBFPとGFPの蛍光を測定が行われ、また各サンプルにつき、3回測定を行ったようです。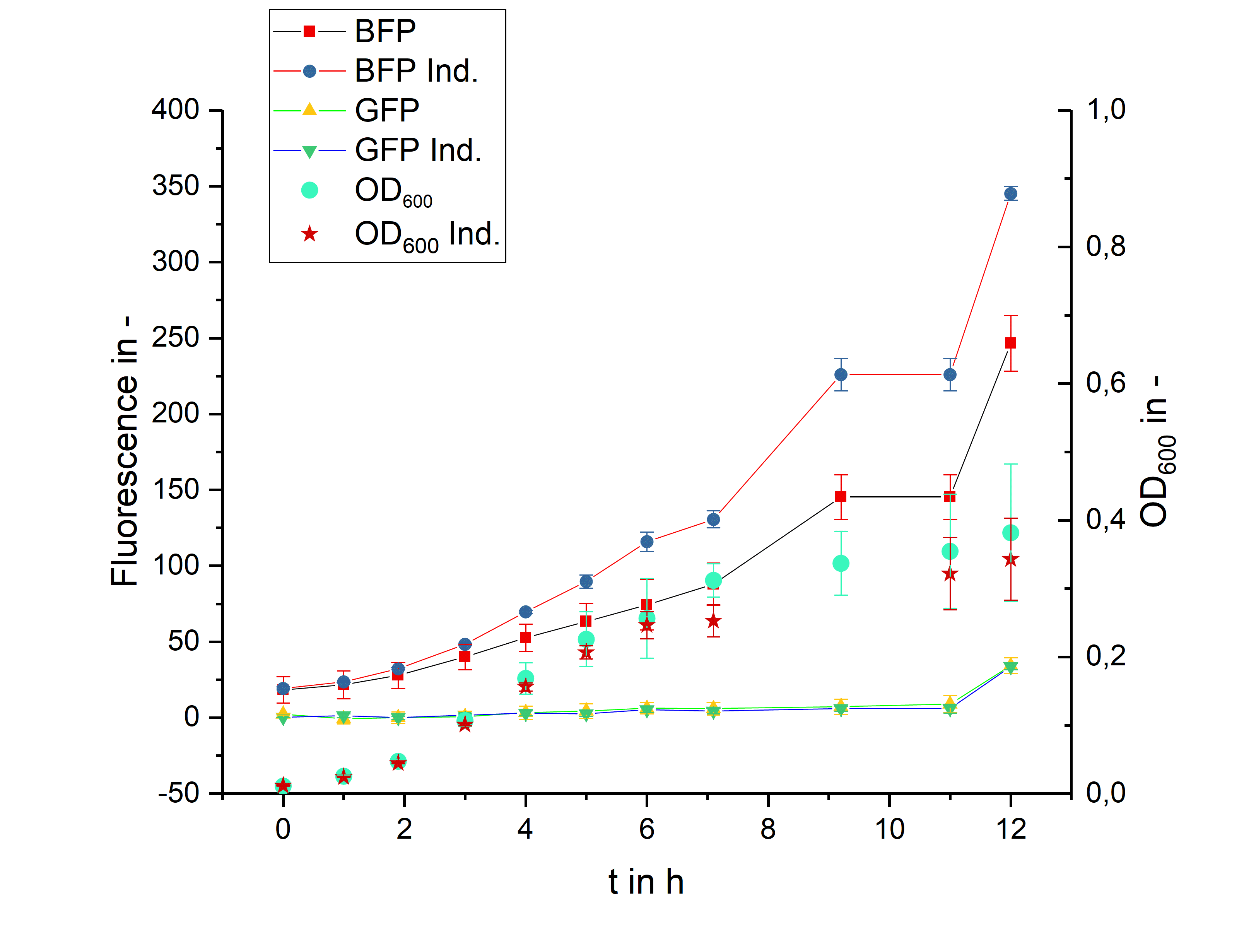
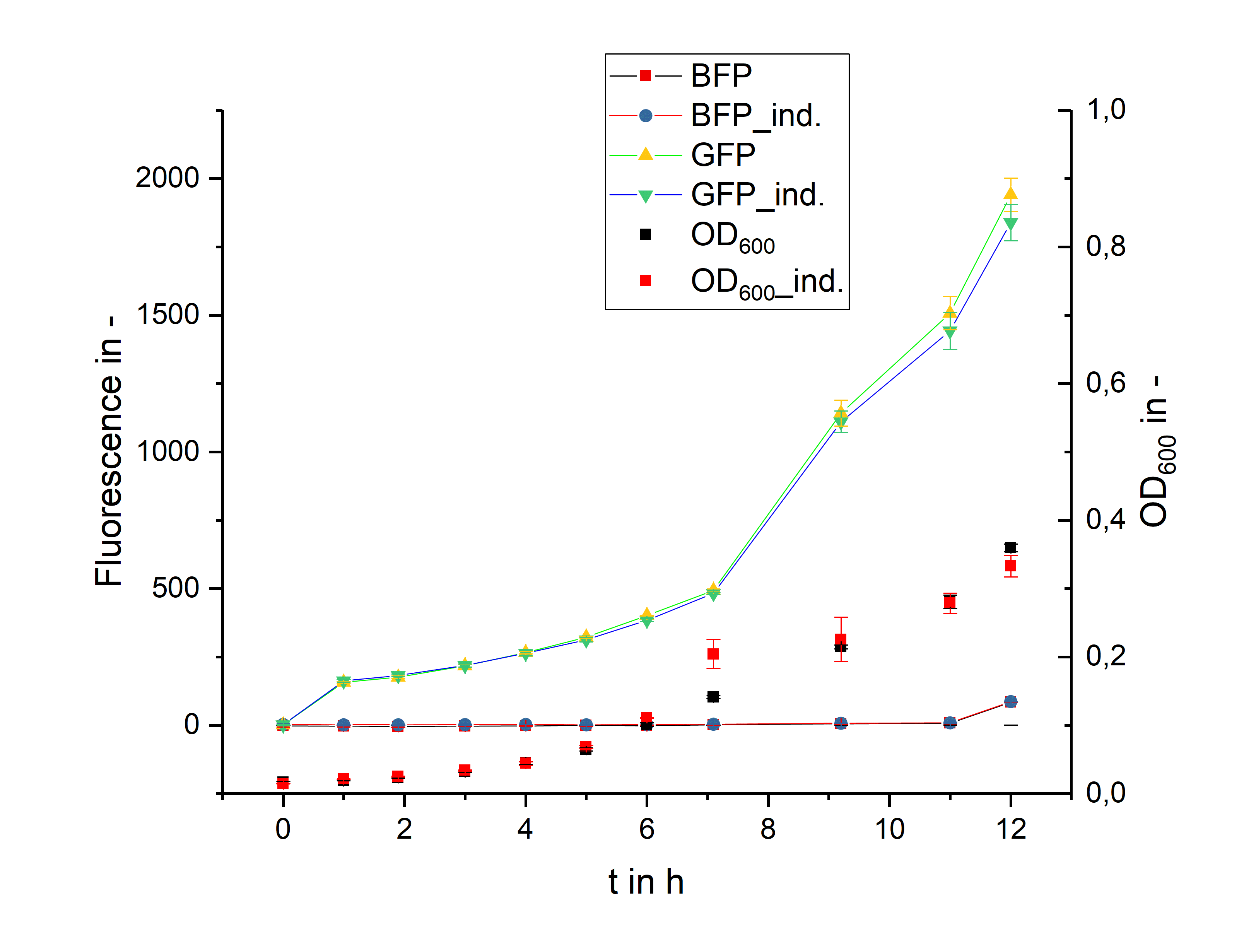
実験結果 上) 1回目, 右) 2回目
出典 : iGEM2018 Bielefeld-CeBiTec nanoFactory Silencing Results https://2018.igem.org/Team:Bielefeld-CeBiTec/siRNA_Results
2つのクローンにおけるそれぞれの実験結果です。左の図のクローンではBFPが発現しているものの、GFPは発現していませんでした。また右の図のクローンではGFPが発現しているものの、BFPは発現していませんでした。
実験の仮説としては、BFPとGFPの両方のタンパク質が発現しますが、両方発現している結果は得られませんでした。
実験結果より、Bielefeld-CeBiTecは、酸素制限培養条件下では実験期間中の細胞の増殖スピードが非常に遅いと主張していますが、ODの条件が揃っていないなど、この主張には疑問が残ります。また、左図の実験結果は、BFPが発現したものの、GFPが発現しなかったというものでしたが、シーケンシングの結果、ターゲットベクターにはGFPは50bpしか挿入されていなかったことがわかり、蛍光シグナルの欠損が確認されたようです。
他にも実験を行っていたようですが、結果がwikiに載っていなかったためその内容は割愛させていただきます。
まとめ
今回の輪読会を通して遺伝子のノックダウンを行うRNAiはiGEMの中で、特定の種に特異的に作用させることができる除草剤や、遺伝子のノックアウトを行うCRISPR/CAS9の代わりとなるように応用することができそうだということがわかりました。また、今回の輪読会を通して、iGEMの合成生物を用いた製品の社会実装を目指すという理念とCRISPRの特許によるライセンス契約に必要性の衝突といった深い部分も垣間見ることができました。